承接上文,讲解 AlphaFold3
Limitations for AlphaFold2
一方面,实验表明 AlphaFold2 模型预测准确性很大程度上依赖于 MSA的序列数,尤其MSA 序列数在30以下时 accuracy 显著下降, 推测MSA 对正确预测蛋白质折叠方式非常重要
另一方面,AlphaFold2 只能预测单个蛋白质的架构,无法准确预测复合物的蛋白质架构, 这也是 AlphaFold3 着手解决的问题之一
最根本性的, AlphaFold2 只能预测蛋白质结构,不能预测 DNA, RNA这类的分子结构,更不能预测复合物结构,而 AlphaFold3解决了这一根本局限,可以接受任意生物分子序列输入并准确给出结构预测
Pipeline
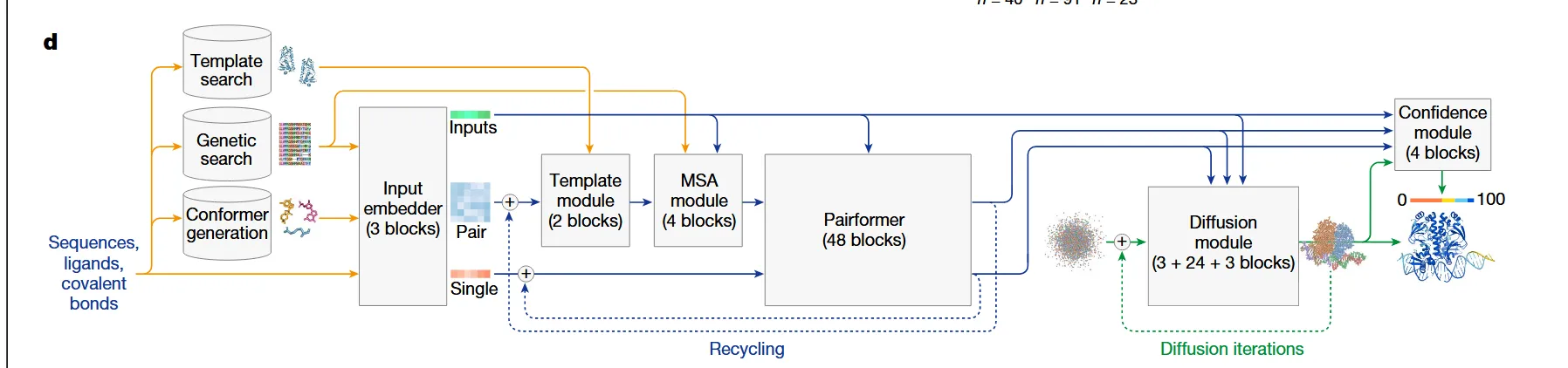
输入
输入比 AlphaFold 2 要明显多样化,可以接受以下输入:
-
序列 (Sequences): 蛋白质、DNA、RNA的序列。
-
配体 (Ligands): 小分子的化学表示法(如SMILES字符串)。
-
共价键 (Covalent bonds): 明确的化学键连接信息。
特征检索
在相关数据库里面完成检索,并最终编码为 Pair(二位关系图)和 Single(一维序列) 这两个输入
由于这部分我们不太关心,因此就不讲解细节了
process trunk
==一个关键变化是把重心放到了 Pair 和 Single 的交换上,很大程度上降低了对 MSA 的依赖程度 ==
1 Template Module
功能是提取同源结构的几何信息,并融入到 Pair 表征中 此部分只包含两个模块
2 MSA Module
大幅简化了MSA信息的提取 使用 Pair-weighed Averaging, 这里并没有提到细节,AI猜测是 根据 Pair 表征 来给每一个同源序列一个权重 这一步之后MSA的信息都被融合进 Pair 中,MSA 不再参与后续信息处理
3 PairFormer
这个模块取到了 AlphaFold2 的 EvoFormer,只输入 Pair 和 Single 表征
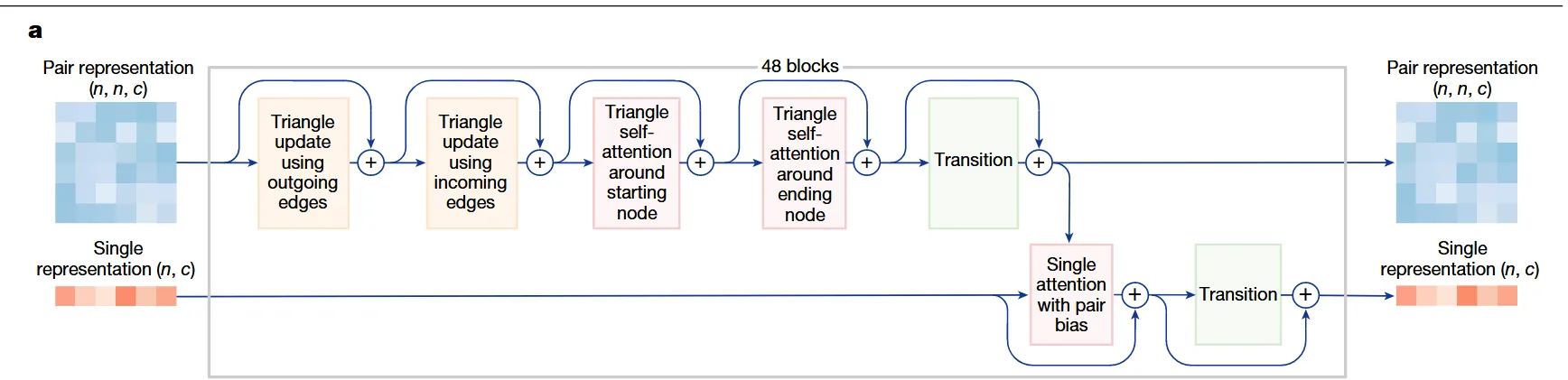 和 AlphaFold 2 相反, 这里先对 Pair 做三角更新, 模块和 AlphaFold 2 一致
和 AlphaFold 2 相反, 这里先对 Pair 做三角更新, 模块和 AlphaFold 2 一致
“带Pair偏置的单体注意力 (Single attention with pair bias)” 的机制传递到Single表征的 。
[!note] 偏置注意力具体实现机制 (AI 提供)
步骤 1: 标准的注意力分数计算
- 首先,对于Single表征中的任意两个元素(token)
i和j,模型会像标准的Transformer一样,通过它们的查询(Query)和键(Key)向量计算一个基础的注意力分数(logit)。这个分数反映了i和j在特征层面的相似性。步骤 2: 从Pair表征中提取“偏置”信息
- 同时,模型会去查询Pair表征这张二维“关系地图”。它找到对应
(i, j)位置的那个格子,并提取出描述这对元素空间关系的特征向量 。步骤 3: 投影并应用偏置
- 提取出的Pair特征向量 会经过一个线性层(Linear Layer)的“投影”,转换成一个单一的数值。
- 这个数值,就是所谓的**“偏置” (bias),它被直接加到**步骤1中计算出的基础注意力分数上。
最终注意力分数(i, j) = 基础注意力分数(i, j) + 来自Pair表征的偏置(i, j)*如果Pair表征显示
i和j在空间上很可能非常接近,那么从 计算出的偏置值就会是一个较大的正数。这会极大地提高i和j之间的最终注意力分数,使得它们在更新自身特征时,会更“关注”对方的信息。
- 反之,如果Pair表征显示
i和j相距很远,偏置值就可能很小甚至是负数,从而降低它们之间的注意力权重。
最后通过一个MLP Transition 来完成处理
Diffusion in structure prediction
这里的Diffusion 和 标准 Diffusion 大体思想是一致的 正向过程就是给生物分子结构增加高斯噪音,把最初的精细结构变成一团 “原子云”
模型学习的是逆向过程:在 Pair 与 Single 的 表征下, 迭代式去噪
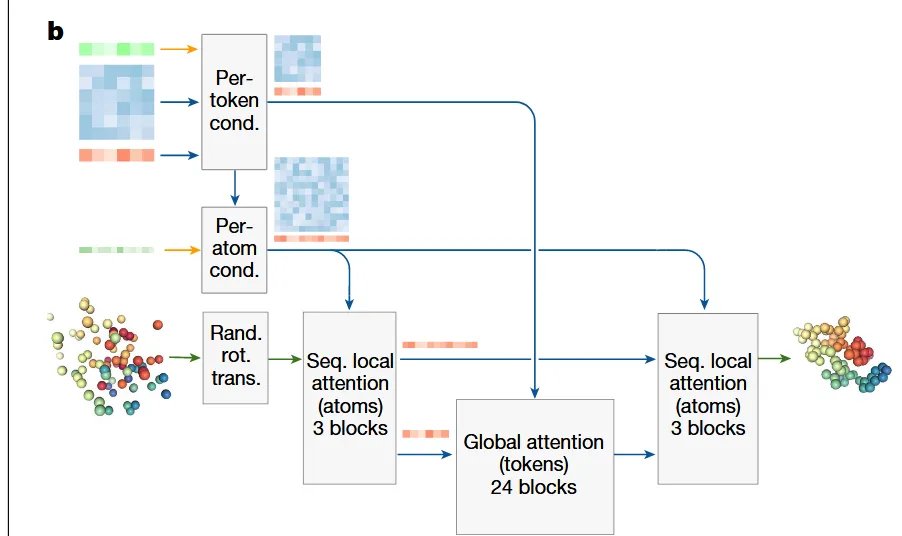 这是模型的单步去噪的网络
这是模型的单步去噪的网络
输入: 带噪声的原子坐标, Pair + Single
处理流程: 类似一个 U-Net, 详细细节看最后AI提供的讲解
[!tip] 生成模型的幻觉处理 众所周知,生成模型很容易产生幻觉(尤其在本来没有固定结构的区域),那么 AF3 是怎么处理这种问题的呢? DeepMind使用了 交叉蒸馏 的策略。它在训练数据中,混入了一部分由 AlphaFold-Multimer v2.3(一个非生成式模型)预测的结构。AF-M在处理无序区时,倾向于生成伸展的、像面条一样的飘带结构。通过学习这些数据,AF3也学会了将无序区预测为类似的伸展结构,而不是错误的紧凑结构,从而大大减少了“幻觉”现象。
置信模块(AI总结)
AlphaFold 3的置信度模块(Confidence Module)是一个至关重要的新组件,其核心用处是对模型自己生成的3D结构进行质量评估,预测其准确性。它不参与结构生成,而是作为一个独立的“质检员”,告诉用户预测结果的哪些部分是可靠的,哪些部分是不可信的。 具体来说,它有以下几个关键作用:
- 逐原子级别的质量评估:置信度模块会预测一个名为 pLDDT (predicted Local Distance Difference Test) 的分数。这是一个0到100之间的值,分配给结构中的每一个原子(或残基),分数越高代表该局部区域的预测结构与真实结构可能越相似。这可以帮助用户识别出高精度折叠的核心区域和可能无序或预测不准的柔性区域。
- 评估相对位置和界面准确性:模块还会预测评估不同部分之间相对位置准确性的指标。
- pAE (Predicted Aligned Error):与AF2一样,它预测如果将预测结构与真实结构的残基
i对齐,那么残基j的位置误差会是多少。这对于评估不同结构域或蛋白链之间堆叠的置信度至关重要]。 - ipTM (interface predicted TM-score):专门用于预测蛋白-蛋白或蛋白-核酸等界面的整体准确性。
- pDE (predicted Distance Error):AF3新增的指标,直接预测原子间距离矩阵的误差。
- pAE (Predicted Aligned Error):与AF2一样,它预测如果将预测结构与真实结构的残基
- 对预测结果进行排序和筛选:在实际使用中,AlphaFold 3通常会运行多次(使用不同的随机种子)以产生多个候选结构 。置信度模块输出的分数(特别是ipTM和pTM)是对这些候选结构进行排序、并选出最佳预测结果的核心依据。例如,在预测抗体-抗原复合物时,研究人员生成了1000个候选结构,并使用ipTM分数来挑选出最可能正确的一个。
实验、局限性与未来方向的总结分析
A. 实验部分总结分析
AlphaFold 3的实验部分旨在系统性地证明其作为一个通用生物分子结构预测模型的卓越性能。
-
蛋白-配体相互作用:
- 基准:PoseBusters,一个包含近期PDB结构的蛋白-配体复合物测试集 [ 757]。
- 结果:在不提供任何真实结构信息(即“盲对接”)的情况下,AF3的准确性(配体RMSD < 2Å的成功率)远超传统的对接工具(如Vina)和其他基于深度学习的盲对接方法(如RoseTTAFold All-Atom) [ 761, 762]。
-
蛋白-核酸相互作用:
- 基准:内部构建的近期PDB评估集,以及CASP15的RNA靶标 [ 593, 768]。
- 结果:AF3预测的蛋白-核酸界面准确性(以iLDDT衡量)远高于专门的核酸预测工具RoseTTAFold2NA [ 764] 。在CASP15 RNA靶标上,其性能也优于最好的纯AI方法,但不及有人类专家干预的方法 [ 768, 769]。
-
共价修饰:
- 基准:内部PDB评估集,涵盖了共价连接的配体、糖基化、以及被修饰的蛋白/核酸残基 [ 772]。
- 结果:AF3能够准确预测这些复杂的共价修饰结构,展示了其处理非标准化学实体的强大能力 [ 772]。
-
蛋白-蛋白相互作用:
- 基准:内部PDB评估集 [ 593]。
- 结果:相较于之前的AlphaFold-Multimer v2.3,AF3在蛋白-蛋白相互作用预测的成功率上(以DockQ > 0.23衡量)有显著提升 [ 782] 。尤其是在抗体-抗原相互作用的预测上,改进尤为明显 [ 783]。
B. 论文中提到的模型局限性 (Model Limitations)
论文作者非常坦诚地指出了当前模型的几个关键局限性:
-
立体化学 (Stereochemistry):
- 手性错误:模型输出的原子结构有时不遵守正确的手性。在PoseBusters测试集上,尽管通过排序惩罚了手性错误,仍有4.4%的违规率。
- 原子冲突 (Clashing):模型偶尔会产生原子在空间上不合理重叠的结构,尤其是在预测大型同源多聚体或大型蛋白-核酸复合物时 。
-
幻觉 (Hallucinations):
- 从AF2的判别式模型转向AF3的生成式Diffusion模型,引入了新的挑战,即在天然无序的区域可能凭空捏造出看似规整的结构(幻觉)。尽管通过交叉蒸馏训练有所缓解,但这个问题依然存在。
-
动力学与多构象 (Dynamics and Conformational States):
- AF3像之前的模型一样,通常只预测一个静态结构,无法捕捉生物分子在溶液中的动态行为或多个构象状态的系综 。
- 在某些情况下,即使输入了配体,模型也可能无法预测出正确的构象状态。例如,E3泛素连接酶在有无配体时应处于不同构象,但AF3对两种情况都只预测了同一种构象 。
-
对挑战性靶标的准确性与算力成本:
- 对于一些非常困难的靶标(如抗体-抗原复合物),为了获得最高精度,可能需要生成大量(例如1000个)的候选预测并进行排序,这会带来巨大的计算成本。
C. 未来方向 (Future Directions)
AF3证明了在一个统一的深度学习框架内,对几乎所有类型的生物分子系统进行高精度结构预测是可能的 [ 989]。未来的方向是继续完善这个通用框架,以应对更广泛、更复杂的生物问题。
-
降低对进化信息的依赖:AF3在抗体等MSA信息稀疏的靶标上取得的巨大进步表明,AlphaFold衍生的方法正在学会直接模拟相互作用的化学和物理原理,而不是仅仅依赖共进化信息 [ 994]。未来的模型可能会进一步降低对MSA的依赖。
-
处理化学多样性:AF3在处理配体方面的成功表明,深度学习框架有能力在没有“蛋白质预测”和“药物对接”人为区分的情况下,处理广阔的化学空间 [ 995]。
-
AI与实验方法的协同进化:未来的结构建模领域的进步,将不仅来源于深度学习的算法突破,也来源于实验结构测定方法(如冷冻电镜)的持续进步 [ 997] 。更多的、高质量的实验数据将为下一代AI模型提供更丰富的训练样本,形成一个计算与实验相互促进的良性循环 [ 998]。
Diffusion模块的单步去噪网络架构(数学讲解) AI 提供
AlphaFold 3的Diffusion模块其本质是一个参数化的分数匹配模型,它被训练来预测并移除加入到真实结构中的噪声。
A. 扩散过程的数学定义
我们首先定义一个正向的、逐步加噪的马尔可夫过程。给定一个真实的原子坐标结构 ,在任意时间步 ( 从0到T),带噪的坐标 可以通过以下公式直接采样得到:
其中:
- 是从标准正态分布 中采样的噪声。
- 是一个预先定义的、随时间步 从1逐渐衰减到0的噪声调度表(noise schedule)。当 时,,此时 (基本无噪声);当 时,,此时 (纯噪声)。
B. 去噪网络 的数学目标
模型的任务是学习一个函数 (一个由权重 参数化的神经网络),该函数能够从任意带噪的结构 中,预测出最可能的原始无噪结构 。这个过程受到来自主干网络(Pairformer)的条件信息 的指导。
这个函数 就是单步去噪网络。在训练时,模型的损失函数通常是预测的 与真实的 之间的均方误差(MSE)。 注意,单步去噪网络在训练中学习的是单步的去噪过程,即如何把一个有一点点噪音的图像、结构变成一个清晰一些的原结构 但在实际预测任务中是从高斯噪声开始,依赖表征的条件,让单步去噪网络多步去噪从而得到标准结构的
C. 单步去噪网络的内部架构 (参考论文图2b)
函数 的内部是一个复杂的多尺度注意力网络,其输入为带噪的原子坐标 和条件信息 ,输出为对原始坐标 的预测。
-
输入嵌入:
- 将输入的原子坐标 嵌入为原子级特征 。
- 将来自主干网络的
Pair和Single表征处理成原子级条件 和令牌级条件 。 - 将时间步 也编码为一个特征向量并融入。
- 令牌级 表征:对于每一个分子(如核苷酸)作为一个token,用一个特征向量表示成的序列 原子级特征:每一个原子维护一个特征向量,尺寸远比 令牌级 表征 要大
-
局部信息处理 (Encoder):
- 网络首先通过3个序列局部注意力模块,在原子层面 上进行计算。这允许模型优先处理局部化学环境。 注意,局部注意力的意思是只和序列顺序上临近原子做处理,可以想象成一个固定大小的窗口(例如 长度 32, 每个原子只和他左右的16个原子计算注意力分数)
-
全局信息处理 (Bottleneck):
- 将精细的原子特征 通过池化(Pooling)操作,聚合成粗粒度的令牌级特征 。
- 然后,通过24个全局注意力模块,在所有令牌之间交换长程信息,推理全局折叠。这里采用的是标准的 QKV 多头注意力
-
局部信息精修 (Decoder):
- 将经过全局处理的令牌特征 通过“去池化”(Unpooling)操作广播回原子级。
- 通常,这里会使用跳跃连接(Skip Connection),将广播回来的特征与第一阶段的原子特征进行拼接,以保留高分辨率的局部信息。
- 最后,再通过3个序列局部注意力模块进行最终的精修。
-
输出头:
- 将最终的原子级特征输入到一个输出头(通常是几个线性层),直接预测出无噪的原子坐标 。
Mini Rollout 方法的进一步解释
Mini Rollout是一种专门为了解决置信度模块训练难题而设计的巧妙策略。
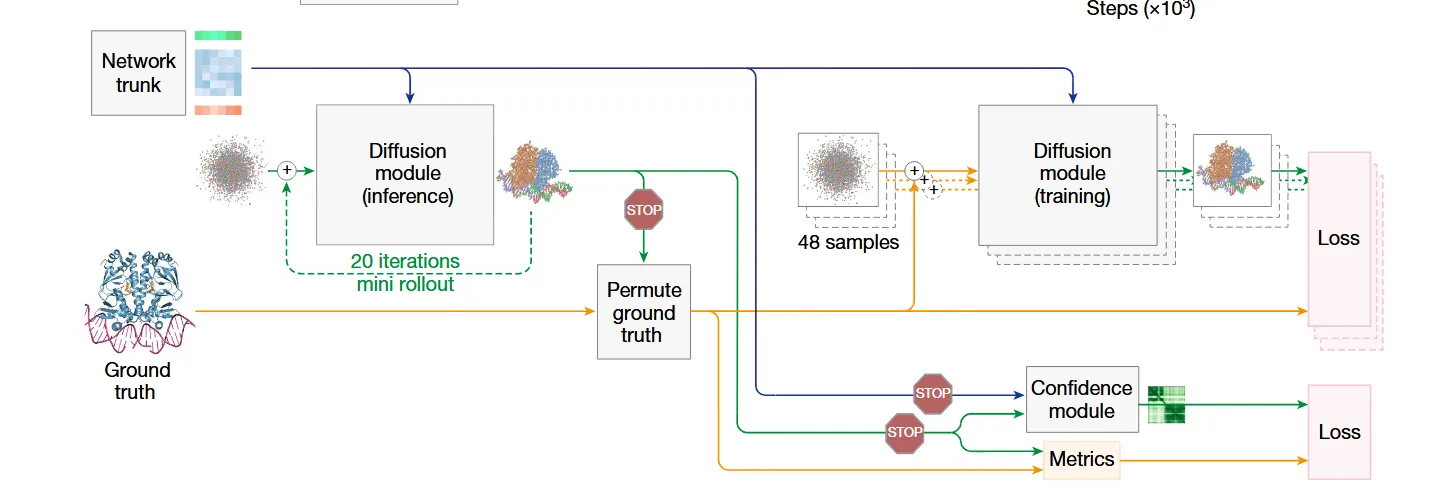
A. 问题的根源
- 置信度模块的目标:它需要预测一个完整生成的结构的准确性。为此,它需要两个东西作为训练数据:一个AI生成的完整结构,以及这个结构相对于真实结构的真实误差。更准确来讲,需要最后生成的”分子结构“
- Diffusion训练的矛盾:标准的高效Diffusion训练过程是单步的。它只学习如何从 (被加过一次随机噪音的样本) 预测 (原样本),而不会在训练中花费大量时间去执行从纯噪声 到最终结构 的完整、多步的生成过程。换句话说,训练单步去噪网络时是不会直接输出完整的”分子结构预测“,只有训练完成在预测的时候才会出现完整的分子结构预测
- 结果:在常规训练步骤中,我们根本没有一个“完整生成的结构”来给置信度模块进行训练。
B. Mini Rollout 的解决方案
为了解决这个矛盾,AlphaFold 3在训练循环中引入了一个并行的、计算开销较低的“模拟推理”过程,这就是Mini Rollout。
其具体流程如下 (参考论文图2c):
-
梯度停止 (Stop Gradient)
- 在一次常规的训练迭代中,主干网络(Pairformer)生成了条件信息 。
- 这些信息兵分两路:一路用于上面描述的单步去噪训练;另一路则进入Mini Rollout流程。
- 进入Mini Rollout的这一路信息被明确地标记为“停止梯度”。这意味着,Mini Rollout过程中产生的任何损失,都不会用来更新主干网络或主Diffusion网络的权重。这保证了主模型的训练不会被这个“旁支任务”所干扰。
-
执行加速的“模拟推理”
- 模型启动一个类似于真实推理的去噪过程,从纯噪声 开始。
- 为了节约计算时间,这个过程是加速的。论文中提到它使用了**“比常规更大的步长”,并且图示中显示它只执行“20次迭代”**(远少于正式推理时的步数)。
- 这个过程的目标不是生成一个完美的结构,而是快速得到一个质量合理的、完整的预测结构 。
-
计算“真实误差”并训练置信度模块
- 一旦 被生成,程序会立刻将它与本次训练样本的真实结构 进行比较,计算出它的真实误差(例如,计算LDDT分数)。
- 同时,将 (或其特征)输入到置信度模块,让它对这个结构的准确性做出预测。
- 最后,通过比较置信度模块的预测值和刚刚算出的真实误差,计算出置信度模块的损失,并用这个损失只更新置信度模块自身的权重。
总结:Mini Rollout就像是在训练主模型(一个学单步解题的学生)的同时,为了训练一个“监考老师”(置信度模块),而进行的模拟小测验。这个测验(Mini Rollout)本身不计入学生的期末成绩(梯度停止),但测验的结果被用来专门训练“监考老师”,让它学会如何准确地评估学生在未来真实考试中的表现。